| Wikipédia : À propos de : | |
| Aide : Contenu : | |
| Aide : Introduction : | |
| Wikipédia : Portail communautaire : | |
| Wikipédia : Assistant de téléchargement de fichiers : | |
| Page d'accueil: |  |
| Aide : Introduction : | |
| Wikipédia : Assistant de téléchargement de fichiers : | |
| Transfert d'ARN : L'ARN de transfert est une molécule adaptatrice composée d'ARN, généralement d'une longueur de 76 à 90 nucléotides, qui sert de lien physique entre l'ARNm et la séquence d'acides aminés des protéines. Pour ce faire, l'ARN de transfert (ARNt) transporte un acide aminé vers la machinerie de synthèse des protéines d'une cellule appelée ribosome. La complémentation d'un codon à 3 nucléotides dans un ARN messager (ARNm) par un anticodon à 3 nucléotides de l'ARNt entraîne une synthèse protéique basée sur le code de l'ARNm. En tant que tels, les ARNt sont un élément nécessaire à la traduction, à la synthèse biologique de nouvelles protéines conformément au code génétique. |  |
| ARN non codant : Un ARN non codant ( ncRNA ) est une molécule d'ARN fonctionnelle qui n'est pas traduite en protéine. La séquence d'ADN à partir de laquelle un ARN fonctionnel non codant est transcrit est souvent appelée gène d'ARN. Les types abondants et fonctionnellement importants d'ARN non codants comprennent les ARN de transfert (ARNt) et les ARN ribosomiques (ARNr), ainsi que les petits ARN tels que les microARN, les siARN, les piARN, les snoARN, les snARN, les exARN, les scaARN et les longs ncARN tels que Xist. et AIR CHAUD. |  |
| RnaG : RnaG est un petit ARN régulateur non codant codé par le plasmide de virulence de Shigella flexneri , une bactérie pathogène à Gram négatif responsable de la dysenterie bacillaire humaine. Il s'agit d'un premier ARN régulateur caractérisé chez S. flexneri . L'ARN mesure 450 nucléotides et contient une région avec une structure secondaire spécifique qui interagit avec l'ARNm icsA et forme un terminateur de transcription. Agissant comme antisens, RnaG est transcrit à partir du brin complémentaire de sa cible, l'ARNm icsA . L'activité de la protéine incA est cruciale pour la propagation du pathogène bactérien dans les cellules hôtes. | 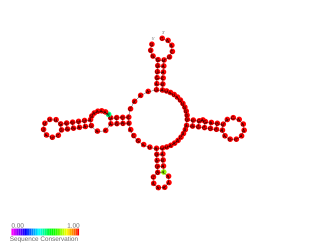 |
| ARN (journal) : RNA est une revue scientifique mensuelle à comité de lecture qui couvre la recherche sur tous les aspects des molécules d'ARN, y compris leurs structures, leur métabolisme, leurs fonctions et leur évolution. La revue a été créée en 1995 et initialement publiée par Cambridge University Press. Depuis 2003, il est publié par Cold Spring Harbor Laboratory Press au nom de la RNA Society. Le rédacteur en chef est Timothy W. Nilsen. | |
| Polyadénylation : La polyadénylation est l'ajout d'une queue poly(A) à un transcrit d'ARN, généralement un ARN messager (ARNm). La queue poly(A) est constituée de plusieurs monophosphates d'adénosine ; en d'autres termes, il s'agit d'une séquence d'ARN qui ne contient que des bases adénine. Chez les eucaryotes, la polyadénylation fait partie du processus qui produit un ARNm mature pour la traduction. Chez de nombreuses bactéries, la queue poly(A) favorise la dégradation de l'ARNm. Il fait donc partie du processus plus vaste d'expression des gènes. | |
| Plafond à cinq premiers : En biologie moléculaire, la coiffe à cinq premiers est un nucléotide spécialement modifié à l'extrémité 5' de certains transcrits primaires tels que l'ARN messager précurseur. Ce processus, connu sous le nom de coiffage de l'ARNm , est hautement régulé et vital dans la création d'ARN messager stable et mature capable de subir une traduction lors de la synthèse des protéines. L'ARNm mitochondrial et l'ARNm chloroplastique ne sont pas coiffés. | |
| ARN polymérase I : L'ARN polymérase 1 est, chez les eucaryotes supérieurs, la polymérase qui transcrit uniquement l'ARN ribosomal, un type d'ARN qui représente plus de 50 % de l'ARN total synthétisé dans une cellule. | |
| ARN polymérase II : L'ARN polymérase II est un complexe multiprotéique qui transcrit l'ADN en précurseurs de l'ARN messager (ARNm) et de la plupart des petits ARN nucléaires (ARNsn) et microARN. C'est l'une des trois enzymes RNAP présentes dans le noyau des cellules eucaryotes. Complexe de 550 kDa de 12 sous-unités, la RNAP II est le type d'ARN polymérase le plus étudié. Un large éventail de facteurs de transcription sont nécessaires pour se lier aux promoteurs de gènes en amont et commencer la transcription. |  |
| ARN polymérase III : Dans les cellules eucaryotes, l'ARN polymérase III est une protéine qui transcrit l'ADN pour synthétiser l'ARN ribosomal 5S, l'ARNt et d'autres petits ARN. | |
| ARN polymérase I : L'ARN polymérase 1 est, chez les eucaryotes supérieurs, la polymérase qui transcrit uniquement l'ARN ribosomal, un type d'ARN qui représente plus de 50 % de l'ARN total synthétisé dans une cellule. | |
| ARN polymérase II : L'ARN polymérase II est un complexe multiprotéique qui transcrit l'ADN en précurseurs de l'ARN messager (ARNm) et de la plupart des petits ARN nucléaires (ARNsn) et microARN. C'est l'une des trois enzymes RNAP présentes dans le noyau des cellules eucaryotes. Complexe de 550 kDa de 12 sous-unités, la RNAP II est le type d'ARN polymérase le plus étudié. Un large éventail de facteurs de transcription sont nécessaires pour se lier aux promoteurs de gènes en amont et commencer la transcription. |  |
| ARN polymérase III : Dans les cellules eucaryotes, l'ARN polymérase III est une protéine qui transcrit l'ADN pour synthétiser l'ARN ribosomal 5S, l'ARNt et d'autres petits ARN. | |
| ARN polymérase : En biologie moléculaire, l'ARN polymérase , ou plus spécifiquement l'ARN polymérase dirigée/dépendante de l'ADN ( DdRP ), est une enzyme qui catalyse les réactions chimiques qui synthétisent l'ARN à partir d'une matrice d'ADN. |  |
| Protéine 44 du motif de liaison à l'ARN : La protéine 44 du motif de liaison à l'ARN est une protéine qui, chez l'homme, est codée par le gène RBM44. |  |
| Protéine de liaison à l'ARN, homologue de fox-1 (c. elegans) 3 : La protéine de liaison à l'ARN, homologue fox-1 3 (Rbfox3) est une protéine qui, chez l'homme, est codée par le gène RBFOX3. Il est lié aux facteurs d'épissage alternatifs Rbfox1 et Rbfox2, mais au lieu de son implication dans l'épissage, il est surtout connu sous le nom de biomarqueur nucléaire NeuN. | |
| Complexe protéique liant la coiffe nucléaire : Le complexe protéique de liaison à la coiffe nucléaire est une protéine de liaison à l'ARN qui se lie à la coiffe 5' du pré-ARNm. La protéine de liaison à la coiffe et à la coiffe nucléaire a de nombreuses fonctions dans la biogenèse de l'ARNm, notamment l'épissage, la formation de l'extrémité 3' en stabilisant l'interaction des machines de traitement de l'extrémité 3', l'exportation nucléaire et la protection des transcrits contre la dégradation des nucléases. Lorsque l'ARN est exporté vers le cytoplasme, le complexe protéique de liaison à la coiffe nucléaire est remplacé par un complexe de liaison à la coiffe cytoplasmique. Le complexe de liaison à la coiffe nucléaire est un hétérodimère fonctionnel composé de Cbc1/Cbc2 chez la levure et de CBC20/CBC80 chez les eucaryotes multicellulaires. Le complexe protéique de liaison à la coiffe nucléaire humaine montre la grande sous-unité, CBC80, composée de 757 résidus d'acides aminés. Sa structure secondaire contient environ soixante pour cent de feuillets hélicoïdaux et un pour cent de feuillet bêta dans le brin. La petite sous-unité CBC20 possède 98 résidus d'acides aminés. Sa structure secondaire contient environ vingt pour cent de feuillets hélicoïdaux et vingt-quatre pour cent de feuillet bêta dans le brin. Le complexe protéique de liaison à la coiffe nucléaire humaine joue un rôle important dans la maturation du pré-ARNm et du petit ARN nucléaire riche en uracile. |  |
| Plafond à cinq premiers : En biologie moléculaire, la coiffe à cinq premiers est un nucléotide spécialement modifié à l'extrémité 5' de certains transcrits primaires tels que l'ARN messager précurseur. Ce processus, connu sous le nom de coiffage de l'ARNm , est hautement régulé et vital dans la création d'ARN messager stable et mature capable de subir une traduction lors de la synthèse des protéines. L'ARNm mitochondrial et l'ARNm chloroplastique ne sont pas coiffés. | |
| Édition d'ARN : L'édition d'ARN est un processus moléculaire par lequel certaines cellules peuvent apporter des modifications discrètes à des séquences nucléotidiques spécifiques au sein d'une molécule d'ARN après qu'elle ait été générée par l'ARN polymérase. Il est présent dans tous les organismes vivants et constitue l'une des propriétés des ARN les plus conservées au cours de l'évolution. L'édition d'ARN peut inclure l'insertion, la suppression et la substitution de bases de nucléotides au sein de la molécule d'ARN. L'édition de l'ARN est relativement rare, les formes courantes de traitement de l'ARN n'étant généralement pas considérées comme une édition. Cela peut affecter l'activité, la localisation ainsi que la stabilité des ARN et a été associé à des maladies humaines. |  |
| Transcription (biologie) : La transcription est le processus de copie d'un segment d'ADN en ARN. On dit que les segments d'ADN transcrits en molécules d'ARN pouvant coder pour des protéines produisent de l'ARN messager (ARNm). D'autres segments d'ADN sont copiés dans des molécules d'ARN appelées ARN non codants (ARNnc). L'ARNm ne représente que 1 à 3 % du total des échantillons d'ARN. Moins de 2 % du génome humain peut être transcrit en ARNm, tandis qu'au moins 80 % de l'ADN génomique des mammifères peut être activement transcrit, la majorité de ces 80 % étant considérée comme étant de l'ARNnc. |  |
| Hélicase : Les hélicases sont une classe d'enzymes considérées comme vitales pour tous les organismes. Leur fonction principale est de décompacter le matériel génétique d'un organisme. Les hélicases sont des protéines motrices qui se déplacent directionnellement le long d'un squelette phosphodiester d'acide nucléique, séparant deux brins d'acide nucléique hybridés, en utilisant l'énergie de l'hydrolyse de l'ATP. Il existe de nombreuses hélicases, représentant la grande variété de processus dans lesquels la séparation des brins doit être catalysée. Environ 1 % des gènes eucaryotes codent pour des hélicases. |  |
| Interférence ARN : L'interférence ARN ( ARNi ) est un processus biologique dans lequel les molécules d'ARN sont impliquées dans la suppression spécifique à une séquence de l'expression des gènes par l'ARN double brin, par répression traductionnelle ou transcriptionnelle. Historiquement, l'ARNi était connu sous d'autres noms, notamment co-suppression , silençage génique post-transcriptionnel (PTGS) et répression . L'étude détaillée de chacun de ces processus apparemment différents a permis de comprendre que l'identité de ces phénomènes était en réalité l'ARNi. Andrew Fire et Craig C. Mello ont partagé le prix Nobel de physiologie ou médecine 2006 pour leurs travaux sur l'ARNi chez le ver nématode Caenorhabditis elegans , qu'ils ont publiés en 1998. Depuis la découverte de l'ARNi et de son potentiel régulateur, il est devenu évident que l'ARNi a un immense potentiel dans la suppression des gènes souhaités. L'ARNi est désormais connu comme étant précis, efficace, stable et meilleur que la thérapie antisens pour la suppression des gènes. L'ARN antisens produit de manière intracellulaire par un vecteur d'expression peut être développé et trouver une utilité en tant que nouveaux agents thérapeutiques. | 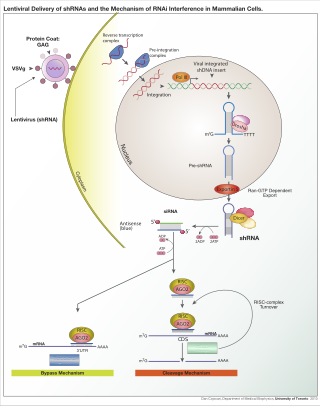 |
| ARN ligase (ATP) : En enzymologie, une ARN ligase (ATP) (EC 6.5.1.3 ) est une enzyme qui catalyse la réaction chimique
| |
| ARN polymérase I : L'ARN polymérase 1 est, chez les eucaryotes supérieurs, la polymérase qui transcrit uniquement l'ARN ribosomal, un type d'ARN qui représente plus de 50 % de l'ARN total synthétisé dans une cellule. | |
| ARN polymérase II : L'ARN polymérase II est un complexe multiprotéique qui transcrit l'ADN en précurseurs de l'ARN messager (ARNm) et de la plupart des petits ARN nucléaires (ARNsn) et microARN. C'est l'une des trois enzymes RNAP présentes dans le noyau des cellules eucaryotes. Complexe de 550 kDa de 12 sous-unités, la RNAP II est le type d'ARN polymérase le plus étudié. Un large éventail de facteurs de transcription sont nécessaires pour se lier aux promoteurs de gènes en amont et commencer la transcription. |  |
| ARN polymérase III : Dans les cellules eucaryotes, l'ARN polymérase III est une protéine qui transcrit l'ADN pour synthétiser l'ARN ribosomal 5S, l'ARNt et d'autres petits ARN. | |
| ARN polymérase I : L'ARN polymérase 1 est, chez les eucaryotes supérieurs, la polymérase qui transcrit uniquement l'ARN ribosomal, un type d'ARN qui représente plus de 50 % de l'ARN total synthétisé dans une cellule. | |
| ARN polymérase II : L'ARN polymérase II est un complexe multiprotéique qui transcrit l'ADN en précurseurs de l'ARN messager (ARNm) et de la plupart des petits ARN nucléaires (ARNsn) et microARN. C'est l'une des trois enzymes RNAP présentes dans le noyau des cellules eucaryotes. Complexe de 550 kDa de 12 sous-unités, la RNAP II est le type d'ARN polymérase le plus étudié. Un large éventail de facteurs de transcription sont nécessaires pour se lier aux promoteurs de gènes en amont et commencer la transcription. |  |
| ARN polymérase III : Dans les cellules eucaryotes, l'ARN polymérase III est une protéine qui transcrit l'ADN pour synthétiser l'ARN ribosomal 5S, l'ARNt et d'autres petits ARN. | |
| ARN polymérase I : L'ARN polymérase 1 est, chez les eucaryotes supérieurs, la polymérase qui transcrit uniquement l'ARN ribosomal, un type d'ARN qui représente plus de 50 % de l'ARN total synthétisé dans une cellule. | |
| ARN polymérase II : L'ARN polymérase II est un complexe multiprotéique qui transcrit l'ADN en précurseurs de l'ARN messager (ARNm) et de la plupart des petits ARN nucléaires (ARNsn) et microARN. C'est l'une des trois enzymes RNAP présentes dans le noyau des cellules eucaryotes. Complexe de 550 kDa de 12 sous-unités, la RNAP II est le type d'ARN polymérase le plus étudié. Un large éventail de facteurs de transcription sont nécessaires pour se lier aux promoteurs de gènes en amont et commencer la transcription. |  |
| ARN polymérase III : Dans les cellules eucaryotes, l'ARN polymérase III est une protéine qui transcrit l'ADN pour synthétiser l'ARN ribosomal 5S, l'ARNt et d'autres petits ARN. | |
| ARN polymérase : En biologie moléculaire, l'ARN polymérase , ou plus spécifiquement l'ARN polymérase dirigée/dépendante de l'ADN ( DdRP ), est une enzyme qui catalyse les réactions chimiques qui synthétisent l'ARN à partir d'une matrice d'ADN. |  |
| Introduction (biologie moléculaire) : Une amorce est un acide nucléique court simple brin utilisé par tous les organismes vivants pour initier la synthèse de l'ADN. Les enzymes ADN polymérase ne sont capables d'ajouter des nucléotides qu'à l'extrémité 3' d'un acide nucléique existant, ce qui nécessite qu'une amorce soit liée à la matrice avant que l'ADN polymérase puisse commencer un brin complémentaire. L'ADN polymérase ajoute des nucléotides après s'être liée à l'amorce d'ARN et synthétise le brin entier. Plus tard, les brins d'ARN doivent être retirés avec précision et remplacés par des nucléotides d'ADN formant une région vide appelée entaille qui est comblée à l'aide d'une enzyme appelée ligase. Le processus d'élimination de l'amorce d'ARN nécessite plusieurs enzymes, telles que Fen1, Lig1 et d'autres, qui fonctionnent en coordination avec l'ADN polymérase, pour assurer l'élimination des nucléotides d'ARN et l'ajout de nucléotides d'ADN. Les organismes vivants utilisent uniquement des amorces d'ARN, tandis que les techniques de laboratoire en biochimie et en biologie moléculaire qui nécessitent une synthèse d'ADN in vitro utilisent généralement des amorces d'ADN, car elles sont plus stables en température. Les amorces peuvent être conçues en laboratoire pour des réactions spécifiques telles que la réaction en chaîne par polymérase (PCR). Lors de la conception d'amorces PCR, certaines mesures spécifiques doivent être prises en compte, comme la température de fusion des amorces et la température d'hybridation de la réaction elle-même. De plus, la séquence de liaison à l'ADN de l'amorce in vitro doit être spécifiquement choisie, ce qui est effectué à l'aide d'une méthode appelée outil de recherche d'alignement local de base (BLAST) qui analyse l'ADN et trouve des régions spécifiques et uniques auxquelles l'amorce doit se lier. |  |
| Sonde d'hybridation : En biologie moléculaire, une sonde d'hybridation (HP) est un fragment d'ADN ou d'ARN d'une longueur généralement de 15 à 10 000 nucléotides qui peut être marqué par radioactivité ou par fluorescence. HP peut être utilisé pour détecter la présence de séquences nucléotidiques dans l'ARN ou l'ADN analysé qui sont complémentaires de la séquence de la sonde. La sonde marquée est d'abord dénaturée (par chauffage ou dans des conditions alcalines telles que l'exposition à l'hydroxyde de sodium) en ADN simple brin (ADNsb), puis hybridée à l'ADNsb cible (Southern blot) ou à l'ARN (northern blot) immobilisé sur une membrane ou dans situ. | |
| Modification post-transcriptionnelle : La modification transcriptionnelle ou modification co-transcriptionnelle est un ensemble de processus biologiques communs à la plupart des cellules eucaryotes par lesquels une transcription primaire d'ARN est chimiquement modifiée suite à la transcription d'un gène pour produire une molécule d'ARN mature et fonctionnelle qui peut ensuite quitter le noyau et effectuer l'une des tâches suivantes. une variété de fonctions différentes dans la cellule. Il existe de nombreux types de modifications post-transcriptionnelles obtenues grâce à diverses classes de mécanismes moléculaires. |  |
| Modification post-transcriptionnelle : La modification transcriptionnelle ou modification co-transcriptionnelle est un ensemble de processus biologiques communs à la plupart des cellules eucaryotes par lesquels une transcription primaire d'ARN est chimiquement modifiée suite à la transcription d'un gène pour produire une molécule d'ARN mature et fonctionnelle qui peut ensuite quitter le noyau et effectuer l'une des tâches suivantes. une variété de fonctions différentes dans la cellule. Il existe de nombreux types de modifications post-transcriptionnelles obtenues grâce à diverses classes de mécanismes moléculaires. |  |
| ARN polymérase ARN-dépendante : L'ARN polymérase dépendante de l'ARN ( RdRp ) ou ARN réplicase est une enzyme qui catalyse la réplication de l'ARN à partir d'une matrice d'ARN. Plus précisément, il catalyse la synthèse du brin d'ARN complémentaire d'une matrice d'ARN donnée. Cela contraste avec les ARN polymérases typiques dépendantes de l'ADN, que tous les organismes utilisent pour catalyser la transcription de l'ARN à partir d'une matrice d'ADN. |  |
| ARN-Seq : RNA-Seq est une technique de séquençage qui utilise le séquençage de nouvelle génération (NGS) pour révéler la présence et la quantité d'ARN dans un échantillon biologique, représentant un instantané agrégé du pool dynamique d'ARN des cellules, également connu sous le nom de transcriptome. |  |
| Épissage d'ARN : L'épissage d'ARN est un processus en biologie moléculaire dans lequel un nouveau transcrit d'ARN messager précurseur (pré-ARNm) est transformé en un ARN messager (ARNm) mature. Cela fonctionne en supprimant tous les introns et en réassemblant les exons. Pour les gènes codés par le noyau, l'épissage se produit dans le noyau pendant ou immédiatement après la transcription. Pour les gènes eucaryotes contenant des introns, un épissage est généralement nécessaire pour créer une molécule d'ARNm pouvant être traduite en protéine. Pour de nombreux introns eucaryotes, l'épissage se produit dans une série de réactions catalysées par le spliceosome, un complexe de petites ribonucléoprotéines nucléaires (snRNP). Il existe des introns auto-épissés, c'est-à-dire des ribozymes capables de catalyser leur propre excision de leur molécule d'ARN parent. Le processus de transcription, d'épissage et de traduction est appelé expression génique, dogme central de la biologie moléculaire. |  |
| Épissage d'ARN : L'épissage d'ARN est un processus en biologie moléculaire dans lequel un nouveau transcrit d'ARN messager précurseur (pré-ARNm) est transformé en un ARN messager (ARNm) mature. Cela fonctionne en supprimant tous les introns et en réassemblant les exons. Pour les gènes codés par le noyau, l'épissage se produit dans le noyau pendant ou immédiatement après la transcription. Pour les gènes eucaryotes contenant des introns, un épissage est généralement nécessaire pour créer une molécule d'ARNm pouvant être traduite en protéine. Pour de nombreux introns eucaryotes, l'épissage se produit dans une série de réactions catalysées par le spliceosome, un complexe de petites ribonucléoprotéines nucléaires (snRNP). Il existe des introns auto-épissés, c'est-à-dire des ribozymes capables de catalyser leur propre excision de leur molécule d'ARN parent. Le processus de transcription, d'épissage et de traduction est appelé expression génique, dogme central de la biologie moléculaire. |  |
| Vaccin à ARNm : Un vaccin à ARNm est un type de vaccin qui utilise une copie d'une molécule appelée ARN messager (ARNm) pour produire une réponse immunitaire. Le vaccin délivre des molécules d'ARNm codant pour l'antigène dans les cellules immunitaires, qui utilisent l'ARNm conçu comme modèle pour construire une protéine étrangère qui serait normalement produite par un agent pathogène ou par une cellule cancéreuse. Ces molécules protéiques stimulent une réponse immunitaire adaptative qui apprend à l'organisme à identifier et à détruire l'agent pathogène ou les cellules cancéreuses correspondantes. L'ARNm est délivré par une co-formulation de l'ARN encapsulé dans des nanoparticules lipidiques qui protègent les brins d'ARN et facilitent leur absorption dans les cellules. |  |
| Classement de Baltimore : La classification de Baltimore est un système utilisé pour classer les virus en fonction de leur mode de synthèse de l'ARN messager (ARNm). En organisant les virus en fonction de leur mode de production d'ARNm, il est possible d'étudier des virus qui se comportent de manière similaire en tant que groupe distinct. Sept groupes de Baltimore sont décrits qui prennent en considération si le génome viral est constitué d'acide désoxyribonucléique (ADN) ou d'acide ribonucléique (ARN), si le génome est simple ou double brin, et si le sens d'un génome à ARN simple brin est décrit. est positif ou négatif. | |
| Classement de Baltimore : La classification de Baltimore est un système utilisé pour classer les virus en fonction de leur mode de synthèse de l'ARN messager (ARNm). En organisant les virus en fonction de leur mode de production d'ARNm, il est possible d'étudier des virus qui se comportent de manière similaire en tant que groupe distinct. Sept groupes de Baltimore sont décrits qui prennent en considération si le génome viral est constitué d'acide désoxyribonucléique (ADN) ou d'acide ribonucléique (ARN), si le génome est simple ou double brin, et si le sens d'un génome à ARN simple brin est décrit. est positif ou négatif. | |
| Classement de Baltimore : La classification de Baltimore est un système utilisé pour classer les virus en fonction de leur mode de synthèse de l'ARN messager (ARNm). En organisant les virus en fonction de leur mode de production d'ARNm, il est possible d'étudier des virus qui se comportent de manière similaire en tant que groupe distinct. Sept groupes de Baltimore sont décrits qui prennent en considération si le génome viral est constitué d'acide désoxyribonucléique (ADN) ou d'acide ribonucléique (ARN), si le génome est simple ou double brin, et si le sens d'un génome à ARN simple brin est décrit. est positif ou négatif. | |
| Le monde de l'ARN : Le monde de l'ARN est une étape hypothétique de l'histoire évolutive de la vie sur Terre, au cours de laquelle les molécules d'ARN auto-réplicatives ont proliféré avant l'évolution de l'ADN et des protéines. Le terme fait également référence à l'hypothèse qui postule l'existence de cette étape. | 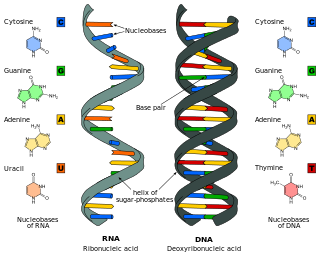 |
| RNAD Broughton Moor : Le RNAD Broughton Moor est un dépôt d'armement de la Royal Naval désaffecté situé entre Great Broughton et Broughton Moor dans le comté de Cumbria, en Angleterre. |  |
| Interférence ARN : L'interférence ARN ( ARNi ) est un processus biologique dans lequel les molécules d'ARN sont impliquées dans la suppression spécifique à une séquence de l'expression des gènes par l'ARN double brin, par répression traductionnelle ou transcriptionnelle. Historiquement, l'ARNi était connu sous d'autres noms, notamment co-suppression , silençage génique post-transcriptionnel (PTGS) et répression . L'étude détaillée de chacun de ces processus apparemment différents a permis de comprendre que l'identité de ces phénomènes était en réalité l'ARNi. Andrew Fire et Craig C. Mello ont partagé le prix Nobel de physiologie ou médecine 2006 pour leurs travaux sur l'ARNi chez le ver nématode Caenorhabditis elegans , qu'ils ont publiés en 1998. Depuis la découverte de l'ARNi et de son potentiel régulateur, il est devenu évident que l'ARNi a un immense potentiel dans la suppression des gènes souhaités. L'ARNi est désormais connu comme étant précis, efficace, stable et meilleur que la thérapie antisens pour la suppression des gènes. L'ARN antisens produit de manière intracellulaire par un vecteur d'expression peut être développé et trouver une utilité en tant que nouveaux agents thérapeutiques. |  |
| RNAL : Reach North Asia Loop ( RNAL ) est un système de câbles de télécommunications sous-marins auto-réparateurs intra-asiatiques de plusieurs térabits, reliant les principales villes d'Asie. | |
| Ribonucléase : La ribonucléase est un type de nucléase qui catalyse la dégradation de l'ARN en composants plus petits. Les ribonucléases peuvent être divisées en endoribonucléases et exoribonucléases et comprennent plusieurs sous-classes au sein des classes d'enzymes EC 2.7 et 3.1. |  |
| Famille des ribonucléases pancréatiques : La famille des ribonucléases pancréatiques est une superfamille d'endonucléases spécifiques de la pyrimidine présentes en grande quantité dans le pancréas de certains mammifères et de certains reptiles. | 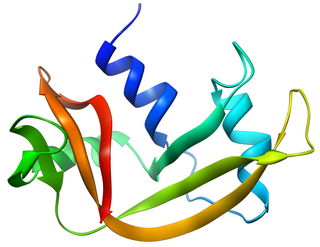 |
| Ribonucléase H : La ribonucléase H est une famille d'enzymes endonucléases non spécifiques à une séquence qui catalysent le clivage de l'ARN dans un substrat ARN/ADN via un mécanisme hydrolytique. Les membres de la famille des RNase H peuvent être trouvés dans presque tous les organismes, des bactéries aux archées en passant par les eucaryotes. |  |
| Ribonucléase P : La ribonucléase P est un type de ribonucléase qui clive l'ARN. La RNase P est unique par rapport aux autres RNases dans la mesure où il s'agit d'un ribozyme – un acide ribonucléique qui agit comme un catalyseur de la même manière qu'une enzyme à base de protéines. Sa fonction est de cliver une séquence d'ARN supplémentaire, ou précurseur, sur les molécules d'ARNt. De plus, la RNase P est l'un des deux ribozymes à renouvellement multiple connus dans la nature, dont la découverte a valu à Sidney Altman et Thomas Cech le prix Nobel de chimie en 1989 : dans les années 1970, Altman a découvert l'existence d'un ARNt précurseur avec des séquences flanquantes et a été le premier à caractériser la RNase P et son activité dans le traitement de la séquence leader 5' de l'ARNt précurseur. Des découvertes récentes révèlent également que la RNase P a une nouvelle fonction. Il a été démontré que la RNase P nucléaire humaine est nécessaire à la transcription normale et efficace de divers petits ARN non codants, tels que les gènes ARNt, ARNr 5S, ARN SRP et snARN U6, qui sont transcrits par l'ARN polymérase III, l'un des trois principaux gènes nucléaires. ARN polymérases dans les cellules humaines. | 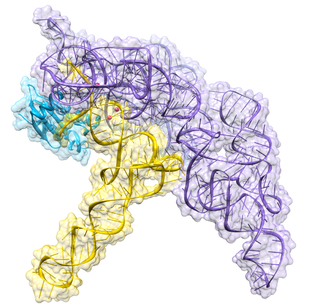 |
| Ribonucléase pancréatique bovine : La ribonucléase pancréatique bovine , également souvent appelée ribonucléase pancréatique bovine A ou simplement RNase A , est une enzyme ribonucléase pancréatique qui clive l'ARN simple brin. La ribonucléase pancréatique bovine est l'un des systèmes modèles classiques de la science des protéines. Deux prix Nobel de chimie ont été décernés pour récompenser des travaux sur la ribonucléase pancréatique bovine : en 1972, le prix a été décerné à Christian Anfinsen pour ses travaux sur le repliement des protéines et à Stanford Moore et William Stein pour leurs travaux sur la relation entre la structure de la protéine et son mécanisme chimique ; en 1984, le prix a été décerné à Robert Bruce Merrifield pour le développement de la synthèse chimique des protéines. |  |
| Ribonucléase V : La ribonucléase V est une enzyme. Cette enzyme catalyse la réaction chimique suivante
| |
| Ribonucléase V1 : La ribonucléase V1 est une enzyme ribonucléase présente dans le venin du cobra caspien. Il clive l'ARN double brin d'une manière non spécifique à une séquence, nécessitant généralement un substrat d'au moins six nucléotides empilés. Comme de nombreuses ribonucléases, l'enzyme nécessite la présence d'ions magnésium pour son activité. |  |
| PH de la RNase : La RNase PH est une nucléotidyltransférase d'ARNt, présente dans les archées et les bactéries, impliquée dans le traitement de l'ARNt. Contrairement aux enzymes hydrolytiques, il s'agit d'une enzyme phosphorolytique, ce qui signifie qu'elle utilise le phosphate inorganique comme réactif pour cliver les liaisons nucléotides-nucléotides, libérant ainsi des nucléotides diphosphates. La structure active des protéines est un complexe homohexamère, constitué de trois dimères de ribonucléase (RNase) PH. La RNase PH a des homologues dans de nombreux autres organismes, appelés protéines de type RNase PH. La partie d'une autre protéine plus grande avec un domaine très similaire à la RNase PH est appelée domaine RNase PH (RPD). |  |
| RNASEH2B : La ribonucléase H2, sous-unité B, est une protéine qui, chez l'homme, est codée par le gène RNASEH2B . La RNase H2 est composée d'une seule sous-unité catalytique (A) et de deux sous-unités non catalytiques et dégrade l'ARN des hybrides ARN : ADN. On pense que la sous-unité B non catalytique de la RNase H2 joue un rôle dans la réplication de l'ADN. | 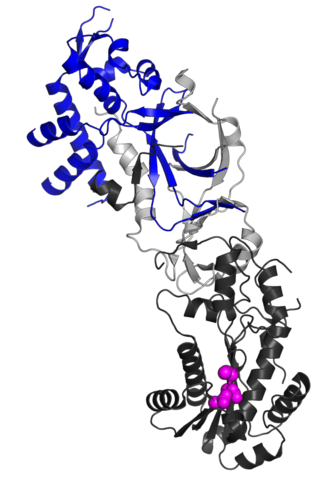 |
| Rythme et blues : Le rythme et le blues , souvent abrégé en R&B ou R'n'B , est un genre de musique populaire originaire des communautés afro-américaines des années 1940. Le terme a été utilisé à l'origine par les maisons de disques pour décrire les enregistrements commercialisés principalement auprès des Afro-Américains, à une époque où « la musique rock basée sur le jazz... [avec un] rythme lourd et insistant » devenait de plus en plus populaire. musique typique des années 1950 aux années 1970, les groupes étaient généralement composés d'un piano, d'une ou deux guitares, d'une basse, d'une batterie, d'un ou plusieurs saxophones et parfois de chanteurs de fond. Les thèmes lyriques du R&B résument souvent l'expérience afro-américaine de la douleur et la quête de liberté et de joie, ainsi que les triomphes et les échecs en termes de relations, d'économie et d'aspirations. |  |
| RnBass : RnBass est un genre musical qui fusionne la musique R&B contemporaine avec un style de production hip hop ou un genre musical, connu sous le nom de musique à cliquet, inventé par DJ Mustard. | |
| RNC : RNC peut faire référence à : | |
| RncO : RncO est un élément régulateur de l'ARN bactérien non codant présent dans la séquence leader du rnc. L'opéron rnc est auto-régulé négativement par la stabilité de la transcription. rnc , le premier gène de l'opéron code pour la RNase III qui clive la longue tige rncO II, conduisant à une dégradation du transcrit et à une réduction de la traduction. Matsunaga et al. ont montré que le clivage de la RNase III peut initier la désintégration du transcrit rnc indépendamment de la traduction du gène rnc. Des travaux supplémentaires ont établi que la structure et la fonction de rncO sont conservées chez Salmonella typhimurium . |  |
| RncO : RncO est un élément régulateur de l'ARN bactérien non codant présent dans la séquence leader du rnc. L'opéron rnc est auto-régulé négativement par la stabilité de la transcription. rnc , le premier gène de l'opéron code pour la RNase III qui clive la longue tige rncO II, conduisant à une dégradation du transcrit et à une réduction de la traduction. Matsunaga et al. ont montré que le clivage de la RNase III peut initier la désintégration du transcrit rnc indépendamment de la traduction du gène rnc. Des travaux supplémentaires ont établi que la structure et la fonction de rncO sont conservées chez Salmonella typhimurium . |  |
| Tour : Rnd peut faire référence à :
| |
| Rédaction du réseau allemand : Le RedaktionsNetzwerk Deutschland ( RND ) est la rédaction commune du groupe allemand Madsack Media, basée à Hanovre. Le plus grand commanditaire de Madsack est la Deutsche Druck- und Verlagsgesellschaft, qui appartient entièrement au Parti social-démocrate allemand (SPD). |  |
| Tour 1 : Rnd1 est une petite protéine G de signalisation et est membre du sous-groupe Rnd de la famille Rho des GTPases. Il est codé par le gène RND1 . |  |
| Tour 2 : Rnd2 est une petite protéine G de signalisation et est membre du sous-groupe Rnd de la famille Rho des GTPases. Il est codé par le gène RND2 . |  |
| Tour 3 : Rnd3 est une petite protéine G de signalisation et est membre du sous-groupe Rnd de la famille Rho des GTPases. Il est codé par le gène RND3 . |  |
| Rnd (GTPase) : Rnd est une sous-classe de la famille Rho de GTPases et comprend :
| |
| Tour : Rnd peut faire référence à :
| |
| Liste des trigraphes en écriture latine : Un certain nombre de trigraphes se trouvent dans l'écriture latine. | |
| Rnd (GTPase) : Rnd est une sous-classe de la famille Rho de GTPases et comprend :
| |
| Jrarat, Kotayk : |  |
| LIER: BIND est une suite de logiciels permettant d'interagir avec le système de noms de domaine (DNS). Son composant le plus important, nommé , remplit les deux principaux rôles de serveur DNS, agissant comme serveur de noms faisant autorité pour les zones DNS et comme résolveur récursif dans le réseau. Depuis 2015, il s'agit du logiciel de serveur de noms de domaine le plus utilisé et constitue le standard de facto sur les systèmes d'exploitation de type Unix. La suite contient également divers outils d'administration tels que nsupdate et dig, ainsi qu'une bibliothèque d'interface de résolution DNS. | |
| Motif d'ARN Rne-II : Le motif d'ARN rne -II est une structure d'ARN conservée identifiée par bioinformatique. On le détecte uniquement chez les espèces classées dans la famille des Pseudomonadaceae, un groupe de gammaprotéobactéries. Les ARN rne -II sont systématiquement situés dans les régions 5' non traduites présumées des gènes qui codent pour la ribonucléase E. L'élément RNase E 5' UTR est une structure d'ARN précédemment identifiée qui se trouve également dans les 5' UTR des gènes de la RNase E. Cependant, ce dernier motif ne se trouve que chez les entérobactéries et les deux motifs ont une structure apparemment indépendante. Compte tenu de leurs différences, il a été émis l'hypothèse que les ARN rne -II remplissent le même rôle fonctionnel que les éléments RNase E 5' UTR, soit de réguler les niveaux de protéines RNase E en agissant comme substrat pour la RNase E. Ainsi, lorsque les concentrations de RNase E sont élevées, elles dégraderont leur propre ARN messager. |  |
| Radio 3 (radio espagnole) : Radio 3 est une station de radio espagnole gratuite détenue et exploitée par Radio Nacional de España (RNE), la division radio du radiodiffuseur public public Radiotelevisión Española (RTVE). Il s'agit de la troisième station de radio de la société et est connue pour diffuser de la musique indépendante, alternative, hip hop et dance. |  |
| Ribonucléase E : La ribonucléase E est une ribonucléase bactérienne qui participe au traitement de l'ARN ribosomal et à la dégradation chimique de l'ARN cellulaire en vrac. | |
| RuneScape : RuneScape est un jeu de rôle en ligne massivement multijoueur (MMORPG) fantastique développé et publié par Jagex, sorti en janvier 2001. RuneScape était à l'origine un jeu par navigateur construit avec le langage de programmation Java ; il a été en grande partie remplacé par un client C++ autonome en 2016. Le jeu a créé plus de 300 millions de comptes et a été reconnu par le Guinness World Records comme le MMORPG gratuit le plus important et le plus mis à jour. | |
| RNF10 : La protéine RING finger 10 est une protéine qui, chez l'homme, est codée par le gène RNF10 . |  |
| Lecture RNF103-CHMP3 : La lecture RNF103-CHMP3 est une protéine qui, chez l'homme, est codée par le gène RNF103-CHMP3. |  |
| Transcription intronique 1 de Rnf216 : Le transcrit intronique 1 RNF216 est une protéine codée par le gène RNF216-IT1 chez l'homme. |  |
| CBL (gène) : Cbl est un gène de mammifère codant pour la protéine CBL qui est une ubiquitine-protéine ligase E3 impliquée dans la signalisation cellulaire et l'ubiquitination des protéines. Des mutations de ce gène ont été impliquées dans un certain nombre de cancers humains, notamment la leucémie myéloïde aiguë. |  |
| Protéine de leucémie promyélocytaire : La protéine de leucémie promyélocytaire ( PML ) est le produit protéique du gène PML . La protéine PML est une protéine suppresseur de tumeur nécessaire à l'assemblage d'un certain nombre de structures nucléaires, appelées corps nucléaires PML, qui se forment parmi la chromatine du noyau cellulaire. Ces corps nucléaires sont présents dans les noyaux des mammifères, à raison d'environ 1 à 30 par noyau cellulaire. Les PML-NB sont connus pour avoir un certain nombre de fonctions cellulaires régulatrices, notamment leur implication dans la mort cellulaire programmée, la stabilité du génome, les effets antiviraux et le contrôle de la division cellulaire. La mutation ou la perte de la PML, et la dérégulation ultérieure de ces processus, ont été impliquées dans divers cancers. |  |
| Sonnerie : Rng ou RNG peut faire référence à : | |
| Rng (algèbre): En mathématiques, et plus particulièrement en algèbre abstraite, un rng est une structure algébrique satisfaisant les mêmes propriétés qu'un anneau, mais sans supposer l'existence d'une identité multiplicative. Le terme rng signifie qu'il s'agit d'un anneau sans i , c'est-à-dire sans l'exigence d'un élément d'identité. | |
| Sonnerie : Rng ou RNG peut faire référence à : | |
| Rng (algèbre): En mathématiques, et plus particulièrement en algèbre abstraite, un rng est une structure algébrique satisfaisant les mêmes propriétés qu'un anneau, mais sans supposer l'existence d'une identité multiplicative. Le terme rng signifie qu'il s'agit d'un anneau sans i , c'est-à-dire sans l'exigence d'un élément d'identité. |
Chủ Nhật, 17 tháng 9, 2023
Wikipedia:About, ,
Đăng ký:
Đăng Nhận xét (Atom)

Không có nhận xét nào:
Đăng nhận xét